Моя программа для обработки данных фрагментного анализа ДНК
По ходу написания PhD, задолбался GeneMapper-ом, его прибитостью гвоздями к венде, чуть меньшую прибитость гвоздями к Ораклу (GeneMapper ID-X 1.7 уже PostgreSQL использует), жручестью, непереносимостью, сильно платностью (16 килобаксов лицензия на 1 человекоместо!). Ну и подумал, что надо посмотреть, а что есть свободного. Нашёл NCBI OSIRIS, но он только для венды и макоси. Есть fatools, есть что-то для R, но не интерактивное ни разу. А у fatools, как по мне, ещё весьма путанный и слабо комментированный код с однобуквенными или около того именами переменных и прочими подобными прелестями.
По итогу, взял я в зубы BioPython, pyqtgraph, SciPy и начал писать свою программу.
Пока что получается неплохо: добился корректной работы с файлами, где реализовано лишь ограниченное подмножество ABIF, с файлами, полученными до стандартизации ABIF (другие смещения, другие названия полей данных...), с файлами HID (получаются на криминалистических капиллярниках ABI 3500 HID, немного отличаются от стандартных ABIF, не особо описаны), определение размера фрагментов с помощью степенных сплайнов или методом наименьших квадратов (мне эту часть математики не преподавали вообще, тип нафига это химикам или биологам. А вот надо! И тем, и другим! Чтоб не заниматься мазохизмом с вычислением площади пиков распечаткой, вырезанием и взвешиванием, а положения измерением линейкой - да, мы так в универе делали в 2013-2014...).
В планах прикрутить автоматическое определение аллелей (биннинг) и импорт STR панелей из CSV файлов.
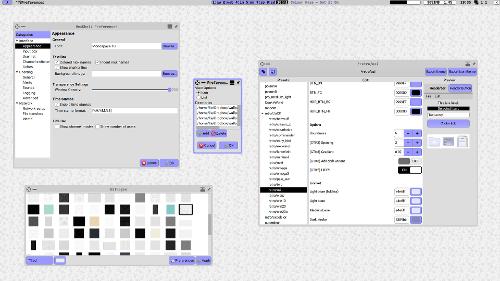
Собственно, на скрине видны данные для аллельного маркера GlobalFiler - рассчитанные размеры в окне программы и предполагаемые с допустимыми отклонениями - в LibreOffice (взято из данных панели GlobalFiler для капиллярника SeqStudio HID). Лицензия программы - AGPL v3. Дистрибутив на скрине openSuSE Tumbleweed, машина - Cisco UCS C240 M3, видеокарта встроенная Matrox с 8Мб видеопамяти, поэтому да, ШГ во все поля.
Ссылка на репозиторий: https://github.com/Dorif/fragalyseqt
И да: кастую  DNA_Seq - давно не видел, может обрадую, что не помер и даже что-то ещё развиваю.)
DNA_Seq - давно не видел, может обрадую, что не помер и даже что-то ещё развиваю.)