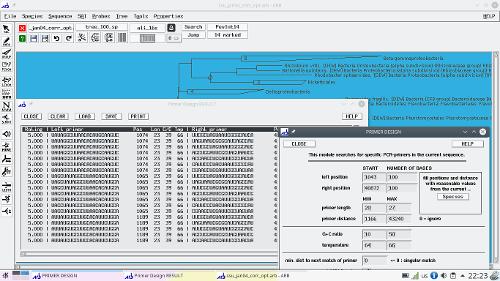
На скриншоте взрыв из прошлого - программа Arb для молекулярной филогенетики. Всего-то 2004го года выпуска. На скриншоте подбор видо-специфических праймеров к генам 16s РНК. Tk жил, Tk жив, Tk будет жить!

Показаны ответы на комментарий. Показать все комментарии.
Ответ на:
комментарий
от Iron_Bug

Ответ на:
комментарий
от Linfan

Ответ на:
комментарий
от Linfan

Ответ на:
комментарий
от DNA_Seq

Ответ на:
комментарий
от Iron_Bug

Ответ на:
комментарий
от Linfan

Ответ на:
комментарий
от DNA_Seq

Ответ на:
комментарий
от Linfan

Ответ на:
комментарий
от DNA_Seq

Вы не можете добавлять комментарии в эту тему. Тема перемещена в архив.
Похожие темы
- Форум Прикладной программист (2008)
- Форум прикладное сапожничество (2008)
- Форум [прикладная фаллометрия] glxgears (2011)
- Форум Прикладной программист (СПб) (2009)
- Форум Прикладной программист (СПб) (2009)
- Форум Прикладной программист (СПб) (2008)
- Галерея Прикладное ониместроение в Blender (2016)
- Форум Требуется прикладной программист (Питер) (2010)
- Форум Вакансия прикладного программиста (Спб) (2010)
- Форум Требуется прикладной программист (Питер) (2010)